交叉验证是保证模型有效的方法,同时也是防止模型过拟合的方法。但在有限的数据集中,交叉验证容易出现一些错误使用。
本文将介绍在使用交叉验证中,常见的一些错误情况,希望读者在阅读后可以避免再次犯错。
什么是交叉验证?
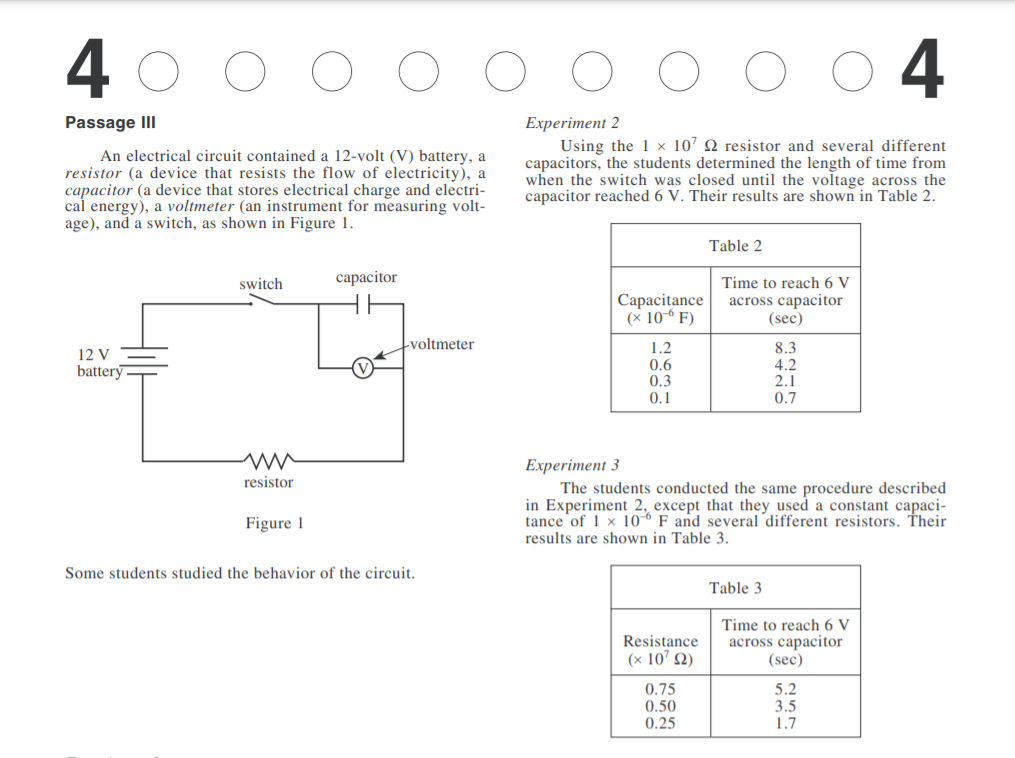
交叉验证(Cross-Validation)是验证模型有效性的方法,具体的实践流程如下:
- 步骤1:数据集划分为K份,其中K-1份作为训练集,剩余1份作为验证集。
- 步骤2:训练集并记录验证集精度。
- 步骤3:将操作上述循环K次。
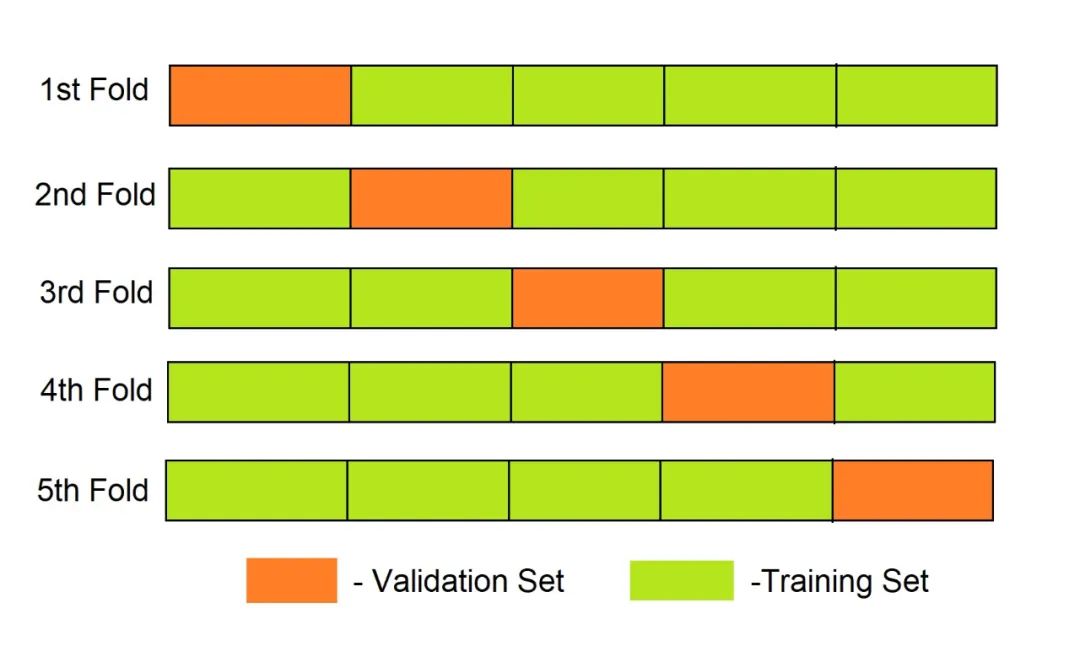
交叉验证与按照比例划分的方法,与如下优点:
- 交叉验证可以验证模型多次,减少了模型误差中的偏差,验证集精度更加可信。
- 交叉验证可以得到多个模型,在测试集上可以进行多次预测,增加预测结果的多样性。
错误1:选择错误的折数
在交叉验证中折数对应K值,是决定了数据集应该划分为多少份,以及模型训练多少次的设置。当然K值需要人工设置,一般设置为5。
K值越小,模型训练次数越少,但模型验证集精度的偏差更大。K值一般不会选择2或者3。K值越大,模型训练的次数越多,需要更多的计算量,但模型验证集的偏差更小。但K值极少数会大于10。
当然选择K值还需要考虑样本的个数,我们希望K值能反应模型验证集精度。另一个可选的方法是尝试多个K值,然后查看模型精度是是否与K值相关。
错误2:数据分布不同
在进行训练与验证时,我们希望训练集和验证集分布保持一致。最直观的分布是标签分布,我们推荐使用StratifiedKFold来代替KFold。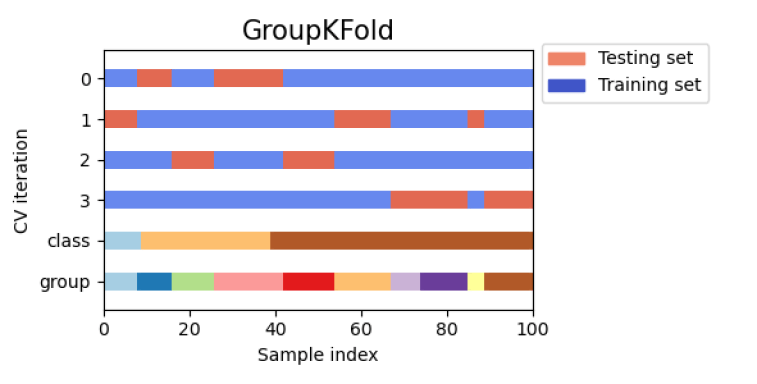 当然也存在一些特殊情况,如果数据样本按照对照组划分,则在划分时也需要考虑分组对照的情况,这里则需要参考StratifiedGroupKfold。
当然也存在一些特殊情况,如果数据样本按照对照组划分,则在划分时也需要考虑分组对照的情况,这里则需要参考StratifiedGroupKfold。 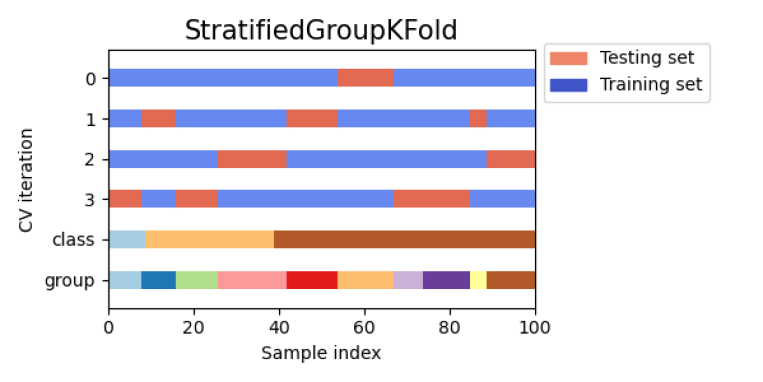
上述划分逻辑只考虑到单个类别情况下的划分,更加复杂的还有数值标签的划分,以及多标签下的情况。
如下演示数值标签的划分,可以将数值进行离散化分箱,然后继续使用StratifiedKFold。
from sklearn.model_selection import StratifiedKFold def create_folds(df, n_grp, n_s=5):
df['Fold'] = -1 skf = StratifiedKFold(n_splits=n_s)
df['grp'] = pd.cut(df.target, n_grp, labels=False)
target = df.grp for fold_no, (t, v) in enumerate(skf.split(target, target)):
df.loc[v, 'Fold'] = fold_no return df
错误3:数据划分后采样
下采样和上采样是解决样本不均衡的常见操作,但如果遇到数据划分,我们是先采样再划分验证集,还是先划分验证集再采样?
这里推荐对验证集不进行任何采样,因为验证集本身是验证模型的精度,用来反应模型的泛化能力。因此验证集数据的分布应该是采样之前的分布。
kfold = KFold(n_splits=n_splits)
scores = [] for train,valid in kfold.split(data):
train_oversampled = oversample_function(train)
score = train_and_validate(train_oversampled,valid)
scores.append(score)
如果采样是必不可少的一步,还是推荐先采样再划分验证集,这样至少训练集和验证集是同分布的。
错误4:过拟合验证集
在我们进行特征工程时,经常会编写如下逻辑的代码:
- 将训练集和测试集拼接一起,进行PCA
- 计算标签编码,然后划分验证集
上述操作都会泄露标签的信息,也就是有隐藏的Leak风险。举一个例子,现在我们对用户进行风险预测,如果按照用户职业进行标签编码,然后再划分验证集,这样会让我们的验证集精度虚高。
正确的做法是先将数据集划分训练集和验证集,在训练集上计算标签编码,然后在验证集上进行映射。 不应该使用验证集进行任何的特征提取和转换过程。
错误5:乱序的时序划分
在时间序列中,数据是按照次序出现的,因此我们在划分验证集时需要考虑时间的先后次序,将验证集划分在训练集之后。当然可以直接参考TimeSeriesSplit划分的逻辑。
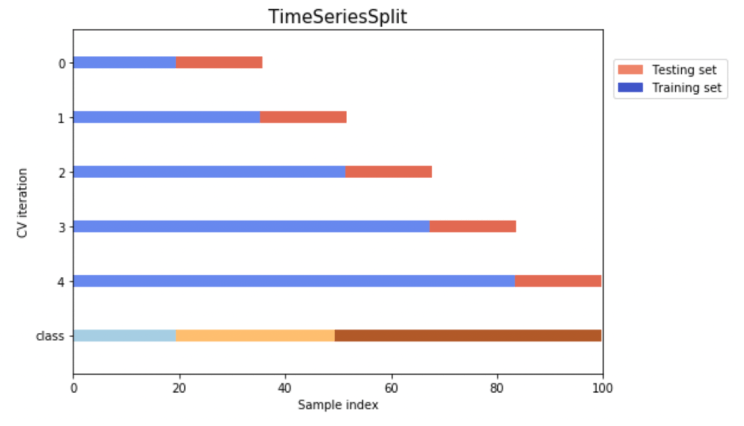
在时序特征中,滞后特征是非常有效的一类。但也经常会泄露验证集信息。滞后特征应该在划分验证集之后,再进行操作。
错误6:数据划分的随机性
虽然数据划分可以得到多个模型,并减少模型验证集的随机性。但数据划分的过程本身还是存在随机性的。
如下代码所示,我们在划分数据集时可以考虑设置随机种子,然后就可以固定数据划分的逻辑。当然也可以训练多次,得到更加稳定的验证集结果。
SEEDS = [1, 2, 3, 4, 5]
ScoreMetric = []
for seed in SEEDS:
seed_all(seed) # 设置所有seed kfold = KFold(n_splits=5, random_state=seed)
scores = []
for train,valid in kfold.split(data):
score = train_and_validate(train,valid)
scores.append(score)
ScoreMetric.append(scores)