文献标题:
Development of Escherichia coli Nissle 1917 derivative by CRISPR/Cas9 and application for gamma-aminobutyric acid (GABA) production in antibiotic-free system
网址:
https://doi.org/10.1016/j.bej.2021.107952
关键词:
Escherichia coli Nissle 1917; Cryptic plasmid; CRISPR/Cas9; Gamma-aminobutyric acid; GABA; Antibiotic-free
主要内容:
一、前言
EcN 作为一种对肠道有益、不致病的大肠杆菌,有很广泛的应用前景。然而,研究发现,野生型 EcN 体内含两种高度稳定的隐性质粒,即 pMUT1(NCBI号MW240712,3.2kb)和 pMUT2(NCBI号CP023342,5.6kb)。由于这两种质粒的存在,导致其作为工程菌的潜力受到限制。不仅异源基因表达受限制,蛋白质表达的效果也不佳,而因为质粒不相容的原因,外源质粒也很难能够转入其中并成功表达。
另一个研究团队发现,利用 CRISPR/Cas 9 技术消除EcN中一个隐性质粒,能够使得外源蛋白的表达强度有所提升,因此,本研究团队打算利用 CRISPR/Cas9 技术消除 pMUT1 和 pMUT2,得到不含质粒的 EcN 衍生菌株 EcNP。
CRISPR/Cas9 是一种基于细菌对入侵者的防御机制的基因组编辑工具,由于其高度准确的 DNA 目标定位特性和成熟的分子工具,已被广泛应用。其中,sgRNA 序列是 CRISPR/Cas9 系统中的关键因素,使 Cas9 酶能够识别和切割目标 DNA。双sgRNA 设计为 Cas9 提供了两个独特的染色体基因缺失靶点,同时也降低了细胞内的 SOS 反应,从而提高了基因编辑能力和效率。
*备注:质粒固化(plasmid curing)是指消除细菌中质粒编码的功能,如抗生素抗性、毒力、芳香化合物的降解等的过程。文献中已经报道了几种质粒固化剂,然而,没有一种质粒固化剂能够消除不同宿主的所有质粒。在质粒稳定或性质损失难以确定的情况下,可以用固化剂处理细菌。这些包括化学和物理试剂,其中一些可以使 DNA 发生突变,特别干扰其复制,或影响细菌细胞的特定结构成分或酶。
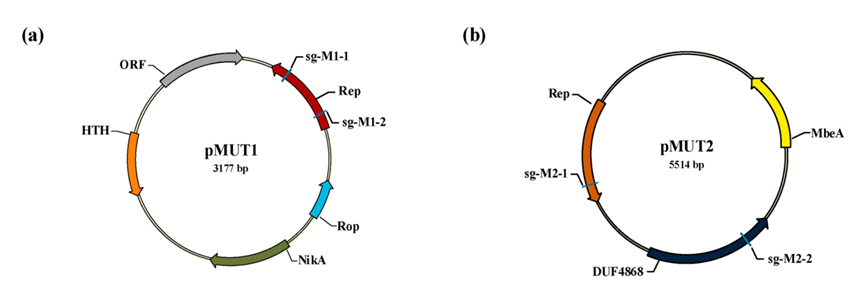
图1:pMUT1 和 pMUT2 的图谱
二、研究思路
研究者们首先发现消除一个 pMUT1 或 pMUT2 可以提高异源基因和蛋白的表达,因此为进行比对,他们在本项研究中先构建了如下的突变体。

图2:本项研究中构建的基础突变体菌株
为了实现质粒的消除,他们在 pCas9-KT 的基础上插入了不同的 sgRNA。这是针对每一个隐质粒设计了一对 sgRNA 序列,其目的是为了将质粒切割成两段,使其难以再次愈合,从而达到质粒消除的作用。
另外,为了研究 EcN 和 EcNP 菌株中异源基因的表达情况,他们又在两种菌株中以不同强度的J系列启动子进行测试。接下来,他们还开发了以 pMUT1 和 pMUT2 为基础的新载体,即 pMT1 和 pMT2,并对其稳定性进行研究,考察其作为一个载体的可能性。最后他们利用 pMT1 和 pMT2,在EcNP中尝试表达 gadB 基因,成功合成了γ-氨基丁酸 (Gamma-aminobutyric acid,GABA)。
本次阅读主要着重于学习如何利用 CRISPR/Cas9 消除 EcN 的两个质粒,以及其相关的验证等一系列操作。
三、材料与方法
为了消除 pMUT1 和 pMUT2,他们构建了一个 pCas9-KT 质粒,并针对不同的用处做出了不同的改变,产生三个衍生物。

图3:pCas9-KT 的衍生质粒
*备注:Km- kanamycin 卡那霉素
sgRNA:small guide RNA 小向导RNA
ori101/repA101ts:一个对温度敏感的复制原点(在37℃下丢失)
载体质粒消除系统的方法如下:
1.首先构建一个含 Cas9 片段、温度敏感起点(ori101/repA101ts)、Km 抗性的 pCas9-KT 质粒。
2.设计 sgRNA 片段,这个 sgRNA 片段两侧含两个 BsaI 酶切位点。sgRNA 的设计是针对不同目标质粒(pMUT1 或 pMUT2)来修改的。

图4:三种 sgRNA 的设计
3. 通过在 Bsa I 位点酶切和酶连,将这些合成的 sgRNA 序列克隆到 pCas9-KT 中,以生成三个质粒 pCK-sgM12、pCK-sgM1 和 pCK-sgM2。
4. 构建好质粒后,将质粒通过化学转化,转入 EcN 中,以无抗 LB 平板涂板,培养过夜。
5. 进行菌落 PCR,随后以 1.8 % 的琼脂糖凝胶验证 PCR 产物。
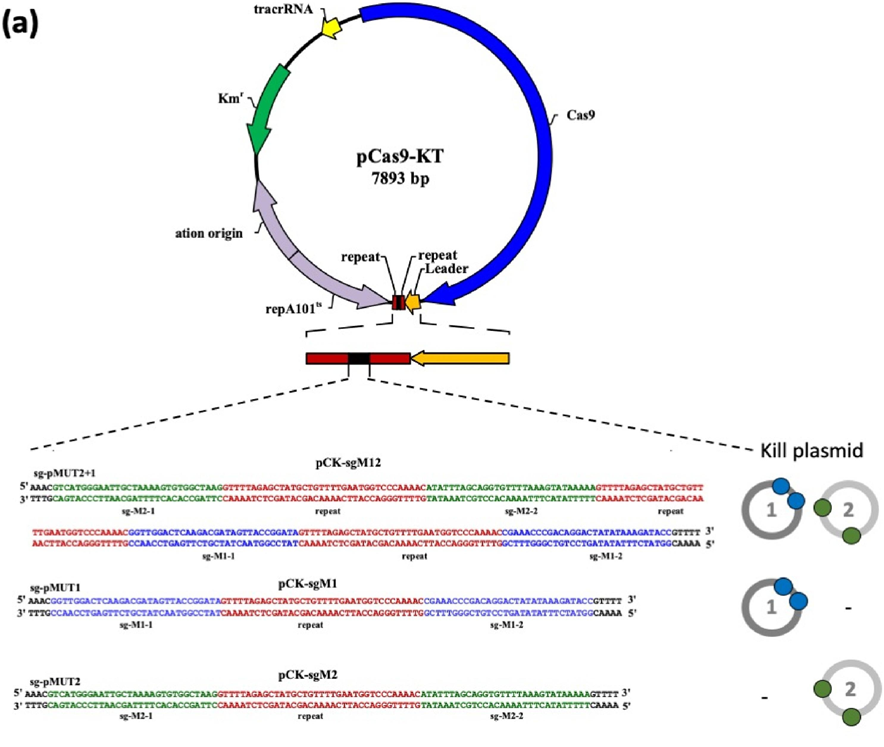
图5:pCas9-KT 与其三个衍生质粒的构建情况
四、结果
菌落PCR跑胶情况如下图所示。
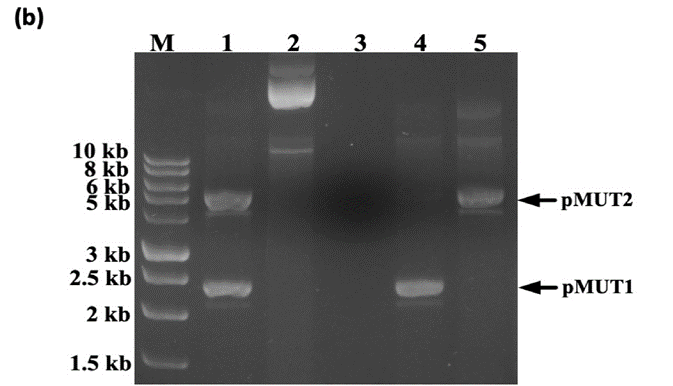
图6
其中,M 为 10kb 的 marker,泳道1为野生EcN PCR结果;泳道为转入 pCas9-KT 的 EcN;泳道 3为 EcNP(即去除了 pMUT1 和 pMUT2);泳道 4为 EcNP1(去除了 pMUT2);泳道 5为 EcNP2(pMUT1 已移除)。箭头表示 pMUT1 和 pMUT2 质粒。
DNA 电泳结果表明,利用研究者们设计的方法,EcN 中的两个隐性质粒均被去除形成了 EcNP,而 EcNP1 和 EcNP2 中分别仅存在 pMUT1 和 pMUT2。这种方法可以同时消除两种隐性质粒,效率高,时间短(小于7天)。
此外,研究者们将不同的J 系列启动子连接 sfGFP 荧光蛋白基因后,导入 EcN 和 EcNP 中表达(J 系列启动子的强度顺序为 J23100 > J23105 > J23114 > J23109),其强度结果测试与 DH5α 中的测试结果一致,且结果表明 EcNP 中 J 系列启动子的表达普遍比 EcN 更强。为了进一步确认质粒的拷贝数(plasmid copy number, PCN)是否影响异源基因的表达情况,将具有不同复制起点的最常见质粒(即pUC、pMB1 和p15A)转化为 EcN 和EcNP 以进行比较。结果表明,EcNP 中的 PCN 高于 EcN 和其他大肠杆菌。
总结来说,一系列测试表明,消除了两个隐性质粒 pMUT1 和 pMUT2 的 EcNP 在异源基因的表达上有更大的优势。
个人评价:
从本篇文献中学习了利用 CRISPR/Cas9 消除 E. coli Nissle 1917 的方法,找到了外源质粒无法转入 E .coli Nissle 1917 的解决办法。后续将以本篇文献为根据,并在师兄指导下完成 E .coli Nissle 1917 的改造,方便后续的其他操作。